Responsable d’équipe : Pr. Gaël Nicolas
 Activités de recherche
Activités de recherche
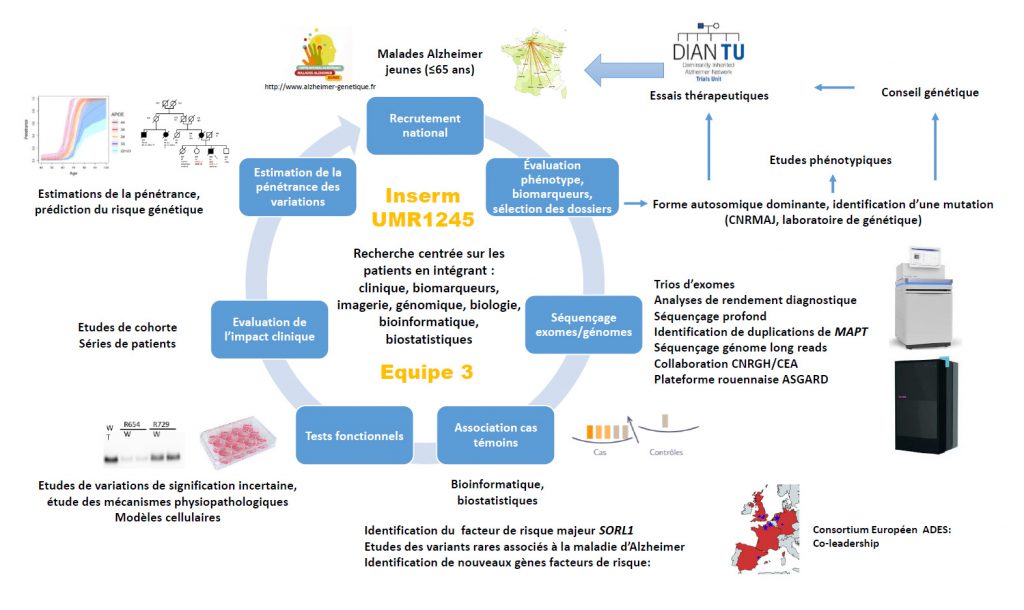
L’équipe Genomics for Brain Disorders (GBD) a pour but d’identifier le déterminisme génétique et les bases moléculaires des maladies neurogénétiques. Les thématiques phares sont la maladie d’Alzheimer et maladies apparentées (angiopathie amyloïde cérébrale, tauopathies, autres démences), les calcifications cérébrales primaires, et les maladies neurodéveloppementales. Appuyée sur un lien étroit avec le CHU de Rouen, le Centre Hospitalier du Rouvray, et un réseau national et international de collaborateurs, l’équipe applique des stratégies de séquençage d’exomes et de génomes à grande échelle et dans de multiples modèles d’études (études cas-témoins, trios, etc.) à partir de prélèvements de patients, et étudie les conséquences des variations identifiées au niveau moléculaire, cellulaire, phénotypique, ainsi que le risque génétique individuel associé aux formes complexes de ces pathologies.
L‘équipe est multidisciplinaire, regroupant des chercheurs, ingénieurs et techniciens experts en génomique, en biologie cellulaire moderne et édition génomique, en bioinformatique et en biostatistiques et des médecins et médecins-chercheurs experts des pathologies neurologiques, psychiatriques, pédiatriques et génétiques.
Le groupe bioinformatique-biostatistiques au sein de l’équipe est en charge des développements méthodologiques nécessaires pour la mise en place des outils d’analyse conformes à l’état de l’art et le développement de stratégies d’analyse innovantes.
Le groupe de biologistes cellulaires est en charge de l’étude des mécanismes physiopathologiques des maladies étudiées, avec un point de vue spécifiquement enrichi sur les variations identifiées chez les patients, en utilisant des technologies de pointe qui ont révolutionné la génomique fonctionnelle : le développement des cellules souches pluripotentes induites (iPSC), leur édition génomique (CRISPR/Cas9) et leur différenciation dans les types cellulaires de choix, en 2D ou en 3D (organoïdes). L’équipe dispose également des outils et de l’expertise pour la modélisation dans le modèle drosophile.
Les différents professionnels travaillent en étroite interaction des patients à la paillasse et vice-versa, dans une vision intégrée et multidisciplinaire des recherches.
L’équipe bénéficie d’un recrutement national pour la plupart des pathologies étudiées. Elle est par exemple adossée au Centre National de Référence Malades Alzheimer Jeunes du CHU de Rouen, en charge du diagnostic et de la recherche génétique pour les malades Alzheimer Jeunes pour tout le territoire national, et elle co-pilote le consortium européen ADES (Alzheimer Disease European Sequencing). Elle est également adossée au laboratoire de génétique moléculaire du CHU de Rouen, reconnu Laboratoire de Biologie Médicale de Référence pour plusieurs pathologies d’intérêt, dont les calcifications cérébrales primaires et les déficiences intellectuelles, incluant le syndrome de Cornelia de Lange et les hydrocéphalies congénitales, et elle travaille en lien direct avec le centre de référence maladies rares (CRMR) Anomalies du Développement et syndromes malformatifs Nord-Ouest, et avec le CRMR maladies neurogénétiques du système nerveux central NEUROGENE, tous deux CRMR constitutifs au CHU de Rouen et travaillant en réseau national.
Les membres de l’équipe sont très impliqués dans la diffusion des connaissances au niveau national, d’une part auprès du grand public, et d’autre part à destination des professionnels et apprenants avec divers degrés d’expertise, à travers des enseignements et conférences de synthèse locaux, nationaux, et internationaux.
 Publications phares
Publications phares
Génétique de la maladie d’Alzheimer et de l’angiopathie amyloïde cérébrale, identification de gènes facteurs de risque à effet modéré à fort par analyse d’exomes cas-témoins, classification clinique des variations facteur de risque, et estimation de leur pénétrance
Nicolas G, Zaréa A, Lacour M, Quenez O, Rousseau S, Richard AC, Bonnevalle A, Schramm C, Olaso R, Sandron F, Boland A, Deleuze JF, Andriuta D, Anthony P, Auria-combe S, Balageas AC, Ballan G, Barbay M, Béjot Y, Belliard S, Benaiteau M, Bennys K, Bombois S, Boutoleau-Bretonnière C, Branger P, Carlier J, Cartz-Piver L, Cassagnaud P, Cec-caldi MP, Chauviré V, Chen Y, Cogez J, Cognat E, Contegal-Callier F, Corneille L, Couratier P, Cretin B, Crinquette C, Dauriat B, Dautricourt S, de la Sayette V, de Liège A, Deffond D, Demurger F, Deramecourt V, Derollez C, Dionet E, Doco Fenzy M, Dumurgier J, Dutray A, Etcharry-Bouyx F, Formaglio M, Gabelle A, Gainche-Salmon A, Godefroy O, Graber M, Gregoire C, Grimaldi S, Gueniat J, Gueriot C, Guillet-Pichon V, Haffen S, Hanta CR, Hardy C, Hautecloque G, Heitz C, Hourregue C, Jonveaux T, Jurici S, Koric L, Krolak-Salmon P, Lagarde J, Lanoiselée HM, Laurens B, Le Ber I, Le Guyader G, Leblanc A, Lebouvier T, Le-vy R, Lippi A, Mackowiak MA, Magnin E, Marelli C, Martinaud O, Maureille A, Migliaccio R, Milongo-Rigal E, Mohr S, Mollion H, Morin A, Nivelle J, Noiray C, Olivieri P, Paquet C, Pariente J, Pasquier F, Perron A, Philippi N, Planche V, Pouclet-Courtemanche H, Rafiq M, Rollin-Sillaire A, Roué-Jagot C, Saracino D, Sarazin M, Sauvée M, Sellal F, Teichmann M, Thauvin C, Thomas Q, Tisserand C, Turpinat C, Van Damme L, Vercruysse O, Villain N, Wa-gemann N, Charbonnier C, Wallon D. Assessment of Mendelian and risk-factor genes in Alzheimer disease: A prospective nationwide clinical utility study and recommendations for genetic screening. Genet Med. 2024 May;26(5):101082. doi: 10.1016/j.gim.2024.101082. Epub 2024 Jan 24. PMID: 38281098.
Holstege* H, Hulsman* M, Charbonnier* C, Grenier-Boley B, Quenez O, Grozeva D, van Rooij JGJ, Sims R, Ahmad S, Amin N, Norsworthy PJ, Dols-Icardo O, Hummerich H, Kawa-lia A, Amouyel P, Beecham GW, Berr C, Bis JC, Boland A, Bossù P, Bouwman F, Bras J, Campion D, Cochran JN, Daniele A, Dartigues JF, Debette S, Deleuze JF, Denning N, DeStefano AL, Farrer LA, Fernández MV, Fox NC, Galimberti D, Genin E, Gille JJP, Le Guen Y, Guerreiro R, Haines JL, Holmes C, Ikram MA, Ikram MK, Jansen IE, Kraaij R, La-throp M, Lemstra AW, Lleó A, Luckcuck L, Mannens MMAM, Marshall R, Martin ER, Ma-sullo C, Mayeux R, Mecocci P, Meggy A, Mol MO, Morgan K, Myers RM, Nacmias B, Naj AC, Napolioni V, Pasquier F, Pastor P, Pericak-Vance MA, Raybould R, Redon R, Reinders MJT, Richard AC, Riedel-Heller SG, Rivadeneira F, Rousseau S, Ryan NS, Saad S, Sanchez-Juan P, Schellenberg GD, Scheltens P, Schott JM, Seripa D, Seshadri S, Sie D, Sistermans EA, Sorbi S, van Spaendonk R, Spalletta G, Tesi N, Tijms B, Uitterlinden AG, van der Lee SJ, Visser PJ, Wagner M, Wallon D, Wang LS, Zarea A, Clarimon J, van Swieten JC, Greicius MD, Yokoyama JS, Cruchaga C, Hardy J, Ramirez A, Mead S, van der Flier WM, van Duijn CM, Williams J, Nicolas** G, Bellenguez** C, Lambert** JC. Exome sequencing identifies rare damaging variants in ATP8B4 and ABCA1 as risk factors for Alzheimer’s dis-ease. Nat Genet. 2022 Dec;54(12):1786-1794. doi: 10.1038/s41588-022-01208-7. Epub 2022 Nov 21. PMID: 36411364; PMCID: PMC9729101.
*cofirst, **colast
Schramm C, Charbonnier C, Zaréa A, Lacour M, Wallon D; CNRMAJ collaborators; Bo-land A, Deleuze JF, Olaso R; ADES consortium; Alarcon F, Campion D, Nuel G, Nicolas G. Penetrance estimation of Alzheimer disease in SORL1 loss-of-function variant carriers using a family-based strategy and stratification by APOE genotypes. Genome Med. 2022 Jun 28;14(1):69. doi: 10.1186/s13073-022-01070-6..
Campion D, Charbonnier C, Nicolas G. SORL1 genetic variants and Alzheimer disease risk: a literature review and meta-analysis of sequencing data. Acta Neuropathol. 2019 Aug;138(2):173-186. doi: 10.1007/s00401-019-01991-4. Epub 2019 Mar 25. PMID: 30911827.
Nicolas G, Charbonnier C, Wallon D, Quenez O, Bellenguez C, Grenier-Boley B, Rousseau S, Richard AC, Rovelet-Lecrux A, Le Guennec K, Bacq D, Garnier JG, Olaso R, Boland A, Meyer V, Deleuze JF, Amouyel P, Munter HM, Bourque G, Lathrop M, Frebourg T, Redon R, Letenneur L, Dartigues JF, Génin E, Lambert JC, Hannequin D, Campion D; CNR-MAJ collaborators. SORL1 rare variants: a major risk factor for familial early-onset Alzheimer’s disease. Mol Psychiatry. 2016 Jun;21(6):831-6. doi: 10.1038/mp.2015.121. Epub 2015 Aug 25. PMID: 26303663.
Grangeon L, Charbonnier C, Zarea A, Rousseau S, Rovelet-Lecrux A, Bendetowicz D, Lemaitre M, Malrain C, Quillard-Muraine M, Cassinari K, Maltete D, Pariente J, Moreaud O, Magnin E, Cretin B, Mackowiak MA, Sillaire AR, Vercelletto M, Dionet E, Felician O, Rod-Olivieri P, Thomas-Antérion C, Godeneche G, Sauvée M, Cartz-Piver L, Le Ber I, Chauvire V, Jonveaux T, Balageas AC, Laquerriere A, Duyckaerts C, Vital A, de Paula AM, Meyronet D, Guyant-Marechal L, Hannequin D, Tournier-Lasserve E, Campion D; CNR-MAJ collabo-rators; Nicolas G, Wallon D. Phenotype and imaging features associated with APP duplications. Alzheimers Res Ther. 2023 May 11;15(1):93. doi: 10.1186/s13195-023-01172-2. PMID: 37170141; PMCID: PMC10173644.
Grangeon L, Cassinari K, Rousseau S, Croisile B, Formaglio M, Moreaud O, Boutonnat J, Le Meur N, Miné M, Coste T, Pipiras E, Tournier-Lasserve E, Rovelet-Lecrux A, Campion D, Wallon D, Nicolas G. Early-Onset Cerebral Amyloid Angiopathy and Alzheimer Disease Related to an APP Locus Triplication. Neurol Genet. 2021 Sep 8;7(5):e609. doi: 10.1212/NXG.0000000000000609. PMID: 34532568; PMCID: PMC8439959.
Caractérisation fonctionnelle des bases moléculaires de la maladie d’Alzheimer ou maladies apparentées dans des modèles cellulaires ou drosophile
Rovelet-Lecrux A, Feuillette S, Miguel L, Schramm C, Pernet S, Quenez O, Ségalas-Milazzo I, Guilhaudis L, Rousseau S, Riou G, Frébourg T, Campion D, Nicolas G, Lecourtois M. Impaired SorLA maturation and trafficking as a new mechanism for SORL1 missense variants in Alzheimer disease. Acta Neuropathol Commun. 2021 Dec 18;9(1):196. doi: 10.1186/s40478-021-01294-4. PMID: 34922638; PMCID: PMC8684260.
Miguel L, Frebourg T, Campion D, Lecourtois M. Moderate Overexpression of Tau in Dro-sophila Exacerbates Amyloid-β-Induced Neuronal Phenotypes and Correlates with Tau Oli-gomerization. J Alzheimers Dis. 2020;74(2):637-647. doi: 10.3233/JAD-190906. PMID: 32065789.
Miguel L, Rovelet-Lecrux A, Feyeux M, Frebourg T, Nassoy P, Campion D, Lecourtois M. Detection of all adult Tau isoforms in a 3D culture model of iPSC-derived neurons. Stem Cell Res. 2019 Oct;40:101541. doi: 10.1016/j.scr.2019.101541. Epub 2019 Aug 23. PMID: 31522011.
Miguel L, Gervais J, Nicolas G, Lecourtois M. SorLA Protective Function Is Restored by Improving SorLA Protein Maturation in a Subset of Alzheimer’s Disease-Associated SORL1 Missense Variants. J Alzheimers Dis. 2023;94(4):1343-1349. doi: 10.3233/JAD-230211. PMID: 37424467.
Feuillette S, Charbonnier C, Frebourg T, Campion D, Lecourtois M. A Connected Net-work of Interacting Proteins Is Involved in Human-Tau Toxicity in Drosophila. Front Neuro-sci. 2020 Feb 11;14:68. doi: 10.3389/fnins.2020.00068. PMID: 32116515; PMCID: PMC7026268.
Miguel L, Frebourg T, Campion D, Lecourtois M. Moderate Overexpression of Tau in Dro-sophila Exacerbates Amyloid-β-Induced Neuronal Phenotypes and Correlates with Tau Oli-gomerization. J Alzheimers Dis. 2020;74(2):637-647. doi: 10.3233/JAD-190906. PMID: 32065789.
Identification d’un nouveau déterminant génétique fort de tauopathie à expression pléiomorphe
Wallon D, Boluda S, Rovelet-Lecrux A, Thierry M, Lagarde J, Miguel L, Lecourtois M, Bonnevalle A, Sarazin M, Bottlaender M, Mula M, Marty S, Nakamura N, Schramm C, Sel-lal F, Jonveaux T, Heitz C, Le Ber I, Epelbaum S, Magnin E, Zarea A, Rousseau S, Quenez O, Hannequin D, Clavaguera F, Campion D, Duyckaerts C, Nicolas G. Clinical and neuro-pathological diversity of tauopathy in MAPT duplication carriers. Acta Neuropathol. 2021 Aug;142(2):259-278. doi: 10.1007/s00401-021-02320-4. Epub 2021 Jun 6. PMID: 34095977.
Le Guennec K, Quenez O, Nicolas G, Wallon D, Rousseau S, Richard AC, Alexander J, Paschou P, Charbonnier C, Bellenguez C, Grenier-Boley B, Lechner D, Bihoreau MT, Olaso R, Boland A, Meyer V, Deleuze JF, Amouyel P, Munter HM, Bourque G, Lathrop M, Frebourg T, Redon R, Letenneur L, Dartigues JF, Martinaud O, Kalev O, Mehrabian S, Traykov L, Ströbel T, Le Ber I, Caroppo P, Epelbaum S, Jonveaux T, Pasquier F, Rollin-Sillaire A, Gé-nin E, Guyant-Maréchal L, Kovacs GG, Lambert JC, Hannequin D, Campion D, Rovelet-Lecrux A. 17q21.31 duplication causes prominent tau-related dementia with increased MAPT expression. Mol Psychiatry. 2017 Aug;22(8):1119-1125. doi: 10.1038/mp.2016.226. Epub 2016 Dec 13. PMID: 27956742.
Miguel L, Rovelet-Lecrux A, Chambon P, Joly-Helas G, Rousseau S, Wallon D, Epel-baum S, Frebourg T, Campion D, Nicolas G, Lecourtois M. Generation of 17q21.31 duplica-tion iPSC-derived neurons as a model for primary tauopathies. Stem Cell Res. 2022 May;61:102762. doi: 10.1016/j.scr.2022.102762. Epub 2022 Mar 22. PMID: 35358831.
Identification ou contribution à l’identification de 4 des 7 gènes de calcifications cérébrales primaires et description du phénotype
Chelban V, Aksnes H, Maroofian R, LaMonica LC, Seabra L, Siggervåg A, Devic P, Shamseldin HE, Vandrovcova J, Murphy D, Richard AC, Quenez O, Bonnevalle A, Zanetti MN, Kaiyrzhanov R, Salpietro V, Efthymiou S, Schottlaender LV, Morsy H, Scardamaglia A, Tariq A, Pagnamenta AT, Pennavaria A, Krogstad LS, Bekkelund ÅK, Caiella A, Glomnes N, Brønstad KM, Tury S, Moreno De Luca A, Boland-Auge A, Olaso R, Deleuze JF, Anheim M, Cretin B, Vona B, Alajlan F, Abdulwahab F, Battini JL, İpek R, Bauer P, Zifarelli G, Gungor S, Kurul SH, Lochmuller H, Da’as SI, Fakhro KA, Gómez-Pascual A, Botía JA, Wood NW, Horvath R, Ernst AM, Rothman JE, McEntagart M, Crow YJ, Alkuraya FS, Nicolas G; SY-NaPS Study Group; Arnesen T, Houlden H. Biallelic NAA60 variants with impaired n-terminal acetylation capacity cause autosomal recessive primary familial brain calcifications. Nat Commun. 2024 Mar 13;15(1):2269. doi: 10.1038/s41467-024-46354-0. PMID: 38480682; PMCID: PMC10937998.
Grangeon L, Wallon D, Charbonnier C, Quenez O, Richard AC, Rousseau S, Budowski C, Lebouvier T, Corbille AG, Vidailhet M, Méneret A, Roze E, Anheim M, Tranchant C, Fa-vrole P, Antoine JC, Defebvre L, Ayrignac X, Labauge P, Pariente J, Clanet M, Maltête D, Rovelet-Lecrux A, Boland A, Deleuze JF; French PFBC study group; Frebourg T, Hannequin D, Campion D, Nicolas G. Biallelic MYORG mutation carriers exhibit primary brain calcification with a distinct phenotype. Brain. 2019 Jun 1;142(6):1573-1586. doi: 10.1093/brain/awz095. PMID: 31009047.
Legati A, Giovannini D, Nicolas G, López-Sánchez U, Quintáns B, Oliveira JR, Sears RL, Ramos EM, Spiteri E, Sobrido MJ, Carracedo Á, Castro-Fernández C, Cubizolle S, Fogel BL, Goizet C, Jen JC, Kirdlarp S, Lang AE, Miedzybrodzka Z, Mitarnun W, Paucar M, Paulson H, Pariente J, Richard AC, Salins NS, Simpson SA, Striano P, Svenningsson P, Tison F, Unni VK, Vanakker O, Wessels MW, Wetchaphanphesat S, Yang M, Boller F, Campion D, Hannequin D, Sitbon M, Geschwind DH, Battini JL, Coppola G. Mutations in XPR1 cause pri-mary familial brain calcification associated with altered phosphate export. Nat Genet. 2015 Jun;47(6):579-81. doi: 10.1038/ng.3289. Epub 2015 May 4. PMID: 25938945; PMCID: PMC4516721.
Nicolas G, Pottier C, Charbonnier C, Guyant-Maréchal L, Le Ber I, Pariente J, Labauge P, Ayrignac X, Defebvre L, Maltête D, Martinaud O, Lefaucheur R, Guillin O, Wallon D, Chaumette B, Rondepierre P, Derache N, Fromager G, Schaeffer S, Krystkowiak P, Verny C, Jurici S, Sauvée M, Vérin M, Lebouvier T, Rouaud O, Thauvin-Robinet C, Rousseau S, Rovelet-Lecrux A, Frebourg T, Campion D, Hannequin D; French IBGC Study Group. Pheno-typic spectrum of probable and genetically-confirmed idiopathic basal ganglia calcification. Brain. 2013 Nov;136(Pt 11):3395-407. doi: 10.1093/brain/awt255. Epub 2013 Sep 24. PMID: 24065723.
Keller A, Westenberger A, Sobrido MJ, García-Murias M, Domingo A, Sears RL, Lemos RR, Ordoñez-Ugalde A, Nicolas G, da Cunha JE, Rushing EJ, Hugelshofer M, Wurnig MC, Kaech A, Reimann R, Lohmann K, Dobričić V, Carracedo A, Petrović I, Miyasaki JM, Aba-kumova I, Mäe MA, Raschperger E, Zatz M, Zschiedrich K, Klepper J, Spiteri E, Prieto JM, Navas I, Preuss M, Dering C, Janković M, Paucar M, Svenningsson P, Saliminejad K, Khorshid HR, Novaković I, Aguzzi A, Boss A, Le Ber I, Defer G, Hannequin D, Kostić VS, Campion D, Geschwind DH, Coppola G, Betsholtz C, Klein C, Oliveira JR. Mutations in the gene encoding PDGF-B cause brain calcifications in humans and mice. Nat Genet. 2013 Sep;45(9):1077-82. doi: 10.1038/ng.2723. Epub 2013 Aug 4. PMID: 23913003.
Nicolas G, Pottier C, Maltête D, Coutant S, Rovelet-Lecrux A, Legallic S, Rousseau S, Vaschalde Y, Guyant-Maréchal L, Augustin J, Martinaud O, Defebvre L, Krystkowiak P, Pariente J, Clanet M, Labauge P, Ayrignac X, Lefaucheur R, Le Ber I, Frebourg T, Hannequin D, Campion D. Mutation of the PDGFRB gene as a cause of idiopathic basal ganglia calcification. Neurology. 2013 Jan 8;80(2):181-7. doi: 10.1212/WNL.0b013e31827ccf34. Epub 2012 Dec 19. PMID: 23255827.
Caractérisation de variations non codantes dans des maladies rares, exemple des calcifications cérébrales et du syndrome de Cornelia de Lange
Rovelet-Lecrux A, Bonnevalle A, Quenez O, Delcroix W, Cassinari K, Richard AC, Boland A, Deleuze JF, Goizet C, Rucar A, Verny C, Nguyen K, Lecourtois M, Nicolas G. Up-stream open reading frame-introducing variants in patients with primary familial brain calcifi-cation. Eur J Hum Genet. 2024 Mar 4. doi: 10.1038/s41431-024-01580-4. Epub ahead of print. PMID: 38433263.
Cassinari K, Rovelet-Lecrux A, Tury S, Quenez O, Richard AC, Charbonnier C, Olaso R, Boland A, Deleuze JF, Besancenot JF, Delpont B, Pouliquen D, Lecoquierre F, Chambon P, Thauvin-Robinet C, Campion D, Frebourg T, Battini JL, Nicolas G. Haploinsufficiency of the Primary Familial Brain Calcification Gene SLC20A2 Mediated by Disruption of a Regula-tory Element. Mov Disord. 2020 Aug;35(8):1336-1345. doi: 10.1002/mds.28090. Epub 2020 Jun 7. PMID: 32506582.
Coursimault J, Cassinari K, Lecoquierre F, Quenez O, Coutant S, Derambure C, Vezain M, Drouot N, Vera G, Schaefer E, Philippe A, Doray B, Lambert L, Ghoumid J, Smol T, Ra-ma M, Legendre M, Lacombe D, Fergelot P, Olaso R, Boland A, Deleuze JF, Goldenberg A, Saugier-Veber P, Nicolas G. Deep intronic NIPBL de novo mutations and differential diag-noses revealed by whole genome and RNA sequencing in Cornelia de Lange syndrome pa-tients. Hum Mutat. 2022 Dec;43(12):1882-1897. doi: 10.1002/humu.24438. Epub 2022 Jul 23. PMID: 35842780.
Coursimault J, Rovelet-Lecrux A, Cassinari K, Brischoux-Boucher E, Saugier-Veber P, Goldenberg A, Lecoquierre F, Drouot N, Richard AC, Vera G, Coutant S, Quenez O, Rolain M, Bonnet C, Bronner M, Lecourtois M, Nicolas G. uORF-introducing variants in the 5’UTR of the NIPBL gene as a cause of Cornelia de Lange syndrome. Hum Mutat. 2022 Sep;43(9):1239-1248. doi: 10.1002/humu.24384. Epub 2022 May 17. PMID: 35446447.
Étude du déterminisme génétique de maladies rares du neurodéveloppement, hydrocéphalies congénitales, troubles du spectre autistique, étude de la contribution des épisignatures au diagnostic des maladies du développement et étude de la récurrence familiale de mutations de novo dans les maladies du développement, développements technologiques et bioinformatiques
Lecoquierre F, Cassinari K, Drouot N, May A, Fourneaux S, Charbonnier F, Derambure C, Coutant S, Saugier-Veber P, Hoischen A, Charbonnier C, Nicolas G. Assessment of paren-tal mosaicism rates in neurodevelopmental disorders caused by apparent de novo pathogenic variants using deep sequencing. Sci Rep. 2024 Mar 4;14(1):5289. doi: 10.1038/s41598-024-53358-9. PMID: 38438430; PMCID: PMC10912112.
Husson T, Lecoquierre F, Nicolas G, Richard AC, Afenjar A, Audebert-Bellanger S, Ba-dens C, Bilan F, Bizaoui V, Boland A, Bonnet-Dupeyron MN, Brischoux-Boucher E, Bonnet C, Bournez M, Boute O, Brunelle P, Caumes R, Charles P, Chassaing N, Chatron N, Cogné B, Colin E, Cormier-Daire V, Dard R, Dauriat B, Delanne J, Deleuze JF, Demurger F, Denommé-Pichon AS, Depienne C, Dieux A, Dubourg C, Edery P, El Chehadeh S, Faivre L, Fergelot P, Fradin M, Garde A, Geneviève D, Gilbert-Dussardier B, Goizet C, Goldenberg A, Gouy E, Guerrot AM, Guimier A, Harzalla I, Héron D, Isidor B, Lacombe D, Le Guillou Horn X, Keren B, Kuechler A, Lacaze E, Lavillaureix A, Lehalle D, Lesca G, Lespinasse J, Levy J, Lyonnet S, Morel G, Jean-Marçais N, Marlin S, Marsili L, Mignot C, Nambot S, Ni-zon M, Olaso R, Pasquier L, Perrin L, Petit F, Pingault V, Piton A, Prieur F, Putoux A, Planes M, Odent S, Quélin C, Quemener-Redon S, Rama M, Rio M, Rossi M, Schaefer E, Rondeau S, Saugier-Veber P, Smol T, Sigaudy S, Touraine R, Mau-Them FT, Trimouille A, Van Gils J, Vanlerberghe C, Vantalon V, Vera G, Vincent M, Ziegler A, Guillin O, Campion D, Charbonnier C. Episignatures in practice: independent evaluation of published episignatures for the molecular diagnostics of ten neurodevelopmental disorders. Eur J Hum Genet. 2024 Feb;32(2):190-199. doi: 10.1038/s41431-023-01474-x. Epub 2023 Oct 23. PMID: 37872275; PMCID: PMC10853222.
Lecoquierre F, Quenez O, Fourneaux S, Coutant S, Vezain M, Rolain M, Drouot N, Boland A, Olaso R, Meyer V, Deleuze JF, Dabbagh D, Gilles I, Gayet C, Saugier-Veber P, Goldenberg A, Guerrot AM, Nicolas G. High diagnostic potential of short and long read genome sequencing with transcriptome analysis in exome-negative developmental disorders. Hum Genet. 2023 Jun;142(6):773-783. doi: 10.1007/s00439-023-02553-1. Epub 2023 Apr 19. PMID: 37076692.
Vezain M, Thauvin-Robinet C, Vial Y, Coutant S, Drunat S, Urtizberea JA, Rolland A, Jacquin-Piques A, Fehrenbach S, Nicolas G, Lecoquierre F, Saugier-Veber P. Retrotrans-poson insertion as a novel mutational cause of spinal muscular atrophy. Hum Genet. 2023 Jan;142(1):125-138. doi: 10.1007/s00439-022-02473-6. Epub 2022 Sep 23. PMID: 36138164.
Coursimault J, Guerrot AM, Morrow MM, Schramm C, Zamora FM, Shanmugham A, Liu S, Zou F, Bilan F, Le Guyader G, Bruel AL, Denommé-Pichon AS, Faivre L, Tran Mau-Them F, Tessarech M, Colin E, El Chehadeh S, Gérard B, Schaefer E, Cogne B, Isidor B, Nizon M, Doummar D, Valence S, Héron D, Keren B, Mignot C, Coutton C, Devillard F, Alaix AS, Amiel J, Colleaux L, Munnich A, Poirier K, Rio M, Rondeau S, Barcia G, Callewaert B, Dheedene A, Kumps C, Vergult S, Menten B, Chung WK, Hernan R, Larson A, Nori K, Stewart S, Wheless J, Kresge C, Pletcher BA, Caumes R, Smol T, Sigaudy S, Coubes C, Helm M, Smith R, Morrison J, Wheeler PG, Kritzer A, Jouret G, Afenjar A, Deleuze JF, Olaso R, Boland A, Poitou C, Frebourg T, Houdayer C, Saugier-Veber P, Nicolas G, Lecoquierre F. MYT1L-associated neurodevelopmental disorder: description of 40 new cases and literature review of clinical and molecular aspects. Hum Genet. 2022 Jan;141(1):65-80. doi: 10.1007/s00439-021-02383-z. Epub 2021 Nov 8. PMID: 34748075.
Quenez O, Cassinari K, Coutant S, Lecoquierre F, Le Guennec K, Rousseau S, Richard AC, Vasseur S, Bouvignies E, Bou J, Lienard G, Manase S, Fourneaux S, Drouot N, Nguyen-Viet V, Vezain M, Chambon P, Joly-Helas G, Le Meur N, Castelain M, Boland A, Deleuze JF; FREX Consortium; Tournier I, Charbonnier F, Kasper E, Bougeard G, Frebourg T, Saugier-Veber P, Baert-Desurmont S, Campion D, Rovelet-Lecrux A, Nicolas G. Detection of copy-number variations from NGS data using read depth information: a diagnostic perfor-mance evaluation. Eur J Hum Genet. 2021 Jan;29(1):99-109. doi: 10.1038/s41431-020-0672-2. Epub 2020 Jun 26. PMID: 32591635; PMCID: PMC7852510.
Marguet F, Vezain M, Marcorelles P, Audebert-Bellanger S, Cassinari K, Drouot N, Chambon P, Gonzalez BJ, Horowitz A, Laquerriere A, Saugier-Veber P. Neuropathological hall-marks of fetal hydrocephalus linked to CCDC88C pathogenic variants. Acta Neuropathol Commun. 2021 Jun 6;9(1):104. doi: 10.1186/s40478-021-01207-5. PMID: 34092257; PMCID: PMC8183048.
Lecoquierre F, Brehin AC, Coutant S, Coursimault J, Bazin A, Finck W, Benoist G, Be-gorre M, Beneteau C, Cailliez D, Chenal P, De Jong M, Degré S, Devisme L, Francannet C, Gérard B, Jeanne C, Joubert M, Journel H, Laurichesse Delmas H, Layet V, Liquier A, Man-gione R, Patrier S, Pelluard F, Petit F, Tillouche N, van Ravenswaaij-Arts C, Frebourg T, Saugier-Veber P, Gruchy N, Nicolas G, Gerard M. Exome sequencing identifies the first genetic determinants of sirenomelia in humans. Hum Mutat. 2020 May;41(5):926-933. doi: 10.1002/humu.23998. Epub 2020 Mar 1. PMID: 32058622.
Vezain M, Lecuyer M, Rubio M, Dupé V, Ratié L, David V, Pasquier L, Odent S, Coutant S, Tournier I, Trestard L, Adle-Biassette H, Vivien D, Frebourg T, Gonzalez BJ, Laquerrière A, Saugier-Veber P. A de novo variant in ADGRL2 suggests a novel mechanism underlying the previously undescribed association of extreme microcephaly with severely reduced sulcation and rhombencephalosynapsis. Acta Neuropathol Commun. 2018 Oct 19;6(1):109. doi: 10.1186/s40478-018-0610-5. PMID: 30340542; PMCID: PMC6195752.
 Membres
Membres
Direction et assistante
Chercheurs – Enseignants-Chercheurs – Cliniciens
Aucune page CV n’a été trouvée. Assurez-vous que les pages CV de cette catégorie sont bien publiées, ou supprimez le bloc.
Post Doctorants
Doctorants
 Contact
Contact
CBG – Cancer and Brain Genomics – UMR1245
UFR Santé
Bâtiment Recherche 1er étage
22, boulevard Gambetta
76183 ROUEN
Secrétariat : 02 35 14 82 80
inserm.u1245@univ-rouen.fr



















